Cell type 찾기 시리즈 : SingleR
cell type annotation은 single-cell data분석에 있어서 가장 기본이 되는 분석 중에 하나지만 아직까지도 그 방법이 정형화되어 있지 않다.
초기에 가장 많이 사용했던 방법은 known marker gene expression을 확인하는 것이었는데, 말 그대로 어떤 cluster에서 알고 있는 cell type의 marker gene의 발현이 보이는지 확인해서 결정하는 방법이다. 실험적으로 가장 오랫동안 사용해 오던 방법으로 믿을만하다고 생각할 수도 있지만,.. 실험적으로 cell type을 구분할때는 surface protein을 많이 확인하기 때문에 우리가 보고 있는 mRNA양과 일치하지 않을수 있다(mRNA필요할때만 사용하고 다 translation돼서 surface로 표시된 후에는 mRNA사라질수도)는 한계가 있고 / Manually 사람이 직접 일일이 확인해가면서 cluster별로 cell type을 지정해야하는데 이는 번거로울뿐 아니라 굉장히 주관적이다. 또한 예상이 가는 Cell type list와 matching되는 marker gene list를 함께 갖고 있어야 이런 분석이 가능하다.
이런 단점들을 보완하기 위해 다양한 tool들이 개발이 되었는데 그 중 하나가 2019년에 발표된 “SingleR”이다.
Aran, Dvir, et al. “Reference-based analysis of lung single-cell sequencing reveals a transitional profibrotic macrophage.” Nature immunology 20.2 (2019): 163-172.
DOI : https://doi.org/10.1038/s41590-018-0276-y
paper download
SingleR: Reference-based annotation
SingleR은 scRNA-seq data를 automatic annotation해주는 tool이다.
이미 cell type을 알고 있는 transcriptomic data인 Reference data와 ( single-cell |
bulk) 지금 cell type을 찾으려고 하는 Query(test) data를 input으로 받아 돌아간다. |
Query data의 cell들은 각각 Reference data와의 similarity를 기준으로 새로운 label을 부여받게 된다. (Markers 별로 Spearman correlation 계산해서 scoring함. 가장 높은 점수 받은 reference data의 label을 부여받음.)
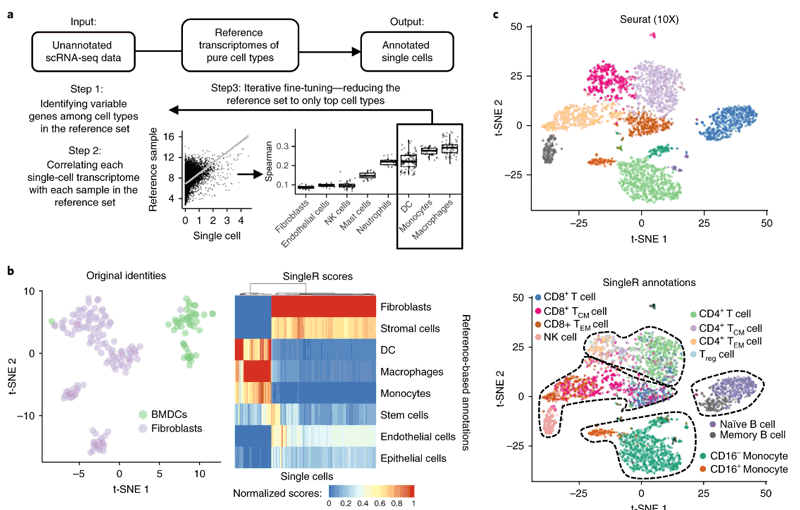 Fig. 1: Reference-based annotation of scRNA-seq
Fig. 1: Reference-based annotation of scRNA-seq
따라서 annotation하려는 Query data와 비슷한 cell type을 포함하고 있는 (이미 annotated된) Reference를 구하면 singleR 분석이 가능하다!! SingleR에는 기본적인 cell type에 대해서는 built-in reference를 제공하고 있기 때문에 바로 실습해보쟈!
SingleR은 R package로도 개발돼서 배포됐을뿐만 아니라 web 버전으로도 제공하고 있다.
Web 버전 SingleR에 들어가면 지금까지 공개된 다양한 scRNA-seq data를 singleR method사용해서 cell type annotation한 결과를 그림(UMAP, tSNE, heatmap plot)과 함께 확인 가능하다.
The SingleR book
이 글에서 소개하는 기능은 singleR의 일부에 불과하다. 더 많은 기능에 대해 알고 싶다면 👆 위에 SingleR book을 참고하길 바란다.
Installation
1
2
#BiocManager 버전 둘중에 하나 골라서 설치
BiocManager::install("SingleR")
Preparation for Reference data
Using built-in references
- celldex
- scRNAseq1
가장 쉬운 방법은 singelR에서 제공해주는 built-in reference를 사용하는 것이다. celldex라는 R package에서 여러가지 referece 를 제공한다.
| Data name | species | description |
|---|---|---|
| BlueprintEncodeData | Human | 259 bulk RNA-seq samples of pure stroma and immune cells as generated and supplied by Blueprint and ENCODE. |
| DatabaseImmuneCellExpressionData | Human | 1561 bulk RNA-seq samples of sorted cell populations from the Database of Immune Cell Expression (DICE). |
| HumanPrimaryCellAtlasData | Human | 713 microarray samples from the Human Primary Cell Atlas (HPCA) (Mabbott et al., 2013). |
| ImmGenData | Mouse | 830 microarray samples of pure mouse immune cells, generated by the Immunologic Genome Project (ImmGen). |
| MonacoImmuneData | Human | 114 bulk RNA-seq samples of sorted |
| immune cell populations that can be found in GSE107011. | ||
| MouseRNAseqData | Mouse | 358 bulk RNA-seq samples of sorted cell |
| populations that can be found at GEO. | ||
| NovershternHematopoieticData | Human | 211 bulk human microarray samples of sorted hematopoietic cell populations that can be found in GSE24759. |
원하는 cell type을 골라잡고 다운 받는다. 인터넷 연결 필요
1
2
library(celldex) #BiocManager::install("celldex")
hpca.se <- celldex::HumanPrimaryCellAtlasData()
🎀서버에서 가끔 말안듣고 다운 안될때
Usage
1
2
3
4
5
6
7
8
9
10
11
library(SingleR)
library(Seurat)
### Load data
# 알아서 자기가 분석하려는 데이터 가져오쟈 (Seurat object기준)
query.data <- readRDS("SeuratObject_afterQC_normalized.rds")
options(future.globals.maxSize = 200000 * 1024^2) #메모리 사이즈 늘려주기
results <- SingleR(test = as.SingleCellExperiment(JE), ref = hpca.se, labels = hpca.se$label.main)
# `lables` : 어떤 label기준으로 prediction할건지 지정. 대부분 main label과 fine label이 있다.
결과 확인
1
2
3
4
5
6
7
8
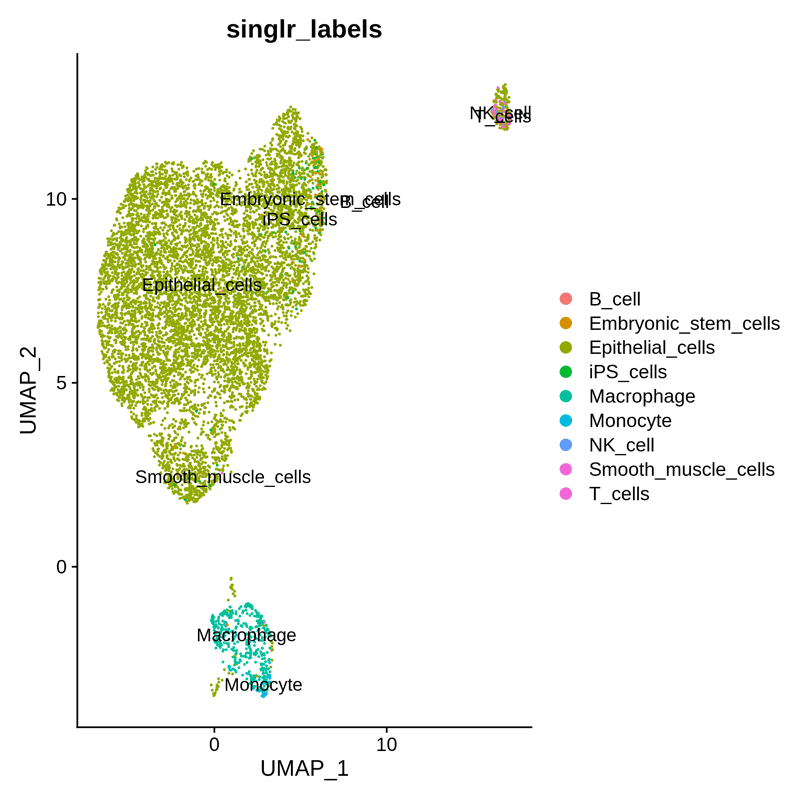
table(results$labels)
B_cell Embryonic_stem_cells Epithelial_cells
2 67 8001
iPS_cells Macrophage Monocyte
95 372 46
NK_cell Smooth_muscle_cells T_cells
10 4 52
그림으로 확인
1
2
3
JE$singlr_labels <- results$labels
DimPlot(JE, reduction = 'umap', group.by = 'singlr_labels', label = TRUE)
ggsave("SingleR.png")
Reference
- Using SingleR to annotate single-cell RNA-seq data
- celldex manual
- https://www.youtube.com/watch?v=LOYQ6zqoApg