Cell type proportion 변화를 표현하는 방법
box plot
지난번에는 찾은 cell type의 비율을 bar plot, pie char로 정리하는 코드를 작성했었는데
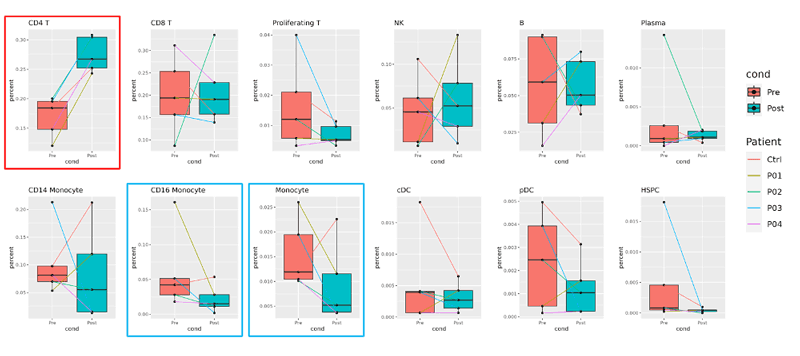
이번에는 cluster별로, condition별로 cell type proportion을 box plot으로 그리고 sample별로 box사이에 line을 이어 그 변화의 흐름을 한눈에 정리해보자.
예시 데이터
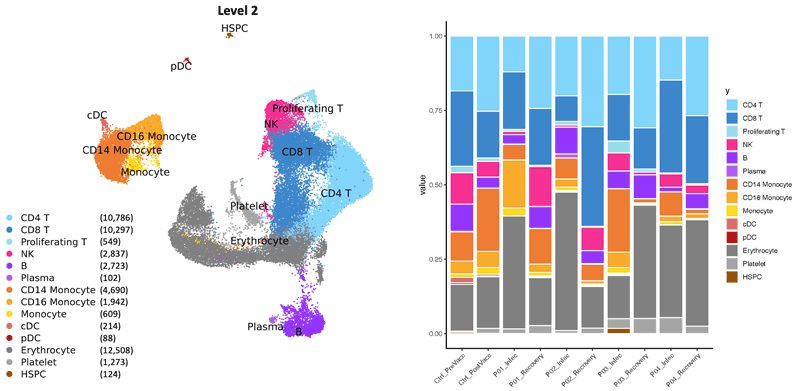
이전에도 썼던 같은 데이터, cell type annotation까지 마친 상태.
하나의 데이터처럼 보이지만, 사실 4명의 다른 환자에서 infected 직후와 recovery 이후에 얻은, 총 8개의 샘플을 합친 결과이다.
샘플별로 cell type proportion을 구하고, infected ⇨ recovery 로 condition변화에 따라 proportion이 어떻게 변화하는지 box plot으로 표현해보자.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
library(ggplot2)
## load the data
GEX.seurOBJ <-readRDS("GEX.seurOBJ.labeled.rds")
# create a dataset
ncluster = length(levels(GEX.seurOBJ$RNA_snn_res.0.8))
Patient <- rep(c('P01','P02','P03','P04'),each=2*ncluster)
Condition <- rep(c("Infec","Recovery"), each=ncluster)
cluster <- levels(GEX.seurOBJ$RNA_snn_res.0.8)
data <- data.frame(Patient,Condition,cluster)
Patient <- rep("Ctrl",2*ncluster)
Condition <- rep(c("PreVacc","PostVacc"), each=ncluster)
data <- rbind(data,data.frame(Patient,Condition,cluster))
data$value <- 0
data$percent <- 0
# cell type proportion
for(k in levels(GEX.seurOBJ$PatientID_re)){
Psub <- subset(GEX.seurOBJ, subset=PatientID_re==k)
if(k=="Ctrl"){
cond_list <- c("PreVacc","PostVacc")
}else{
cond_list <- c("Infec","Recovery")
}
for(i in cond_list){
Csub <- subset(Psub, subset=Condition==i)
total <- ncol(Csub)
for(j in levels(GEX.seurOBJ$RNA_snn_res.0.8)){
data[data$Patient==k & data$Condition==i & data$cluster==j,"value"] <- table(Csub$RNA_snn_res.0.8)[j]
data[data$Patient==k & data$Condition==i & data$cluster==j,"percent"] <- data[data$Patient==k & data$Condition==i & data$cluster==j,"value"]/total
}}}
#write.table(data, "ClusterCounts_by_sample.txt", sep='\t')
# Box plot with line
Infec.data <- data[data$Condition%in%c("Infec","Recovery"),]
Vacc.data <- data[data$Condition%in%c("PreVacc","PostVacc"),]
for(i in unique(Infec.data$cluster)){
ggplot(Infec.data[Infec.data$cluster==i,], aes(Condition, percent, fill=Condition)) +
geom_boxplot()+ geom_point()+ geom_line(aes(group=Patient, col=Patient)) +
labs(title = i,size = 30, face = "bold") +theme_classic() + theme(legend.position="none")
ggsave(paste0("proportion_Infec_cluster",i,".png"),width=2,height=4)
}
for(i in unique(Vacc.data$cluster)){
ggplot(Vacc.data[Vacc.data$cluster==i,], aes(Condition, percent, fill=Condition)) +
geom_boxplot()+ geom_point()+ geom_line(aes(group=Patient, col=Patient)) +
labs(title = i,size = 30, face = "bold") +theme_classic() + theme(legend.position="none")
ggsave(paste0("proportion_Vacc_cluster",i,".png"),width=2,height=4)
}
results
끝
This post is licensed under
CC BY 4.0
by the author.