Cell type proportion을 비교하는 방법: Odd Ratio
이전에도 몇번 cell type identification이후에 proportion을 가시화하고 비교하는 방법들을 몇가지 정리했었는데..
👉 cell type proportion barplot으로 표현하기 포스트
👉 Cell type proportion 변화를 표현하는 방법: lined boxplot 포스트
👉 cell type proportion pie chart로 표현하기 포스트
오늘은 만약에 샘플들 사이에 condition이 있다면, Odd Ratio를 사용해서 두가지 condition사이의 (Ctrl 대비 Treated 사이등) cell type proportion이 있는지, 확인하고 그림으로 표현해서 보여줄 수 있는 방법을 정리해보려함.
Odd Ratio
Odd Ratio란 어떤 특정 사건이 일어날 확률과 그 사건이 일어나지 않을 확률의 비율. 간단히 말해서, 어떤 사건이 발생할 ‘기회’를 수치로 나타낸 것이다.
\[Odd Ratio (OR)= \frac{사건이 발생하지 않을 확률}{사건이 발생할 확률}\]우리는 두가지 Condition에 대해서 OR을 구하고 싶으니까 두 그룹 간의 발생확률의 비율을 구하면 된다. 먼저 그룹별로 사건이 발생할 확률과 그렇지 않을 확률의 비율인 Odds를 구하고, 두 Odds의 비율을 계산하면 Odd Ratio가 됨
\[Odds(A) = \frac{P(A)}{(1 - P(A))}\] \[Odds(B) = \frac{P(B)}{(1 - P(B))}\] \[Odds Ratio = \frac{Odds(A)}{Odds(B)}\]이 비율이 1보다 크면 조건 A에서 사건이 발생할 확률이 조건 B보다 높음을 의미한다. 반대로, 이 비율이 1보다 작으면 조건 B에서 사건이 발생할 확률이 조건 A보다 높다는 것. 이게 통계적 유의성을 갖는지는 다른 test를 통해서 P-val 또는 FDR을 구해서 증명하도록 하자.
Single-Cell data에서의 Odd Ratio 계산
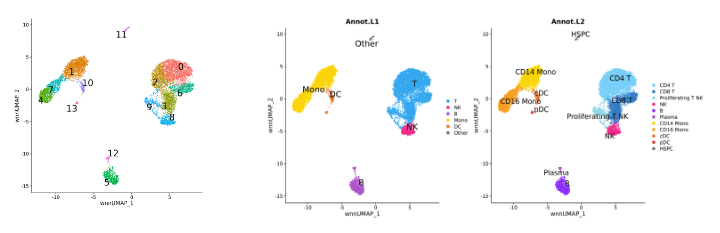
예를들어 내 data가 어떤 바이러스에 감염된 집단에서 얻은 샘플(Infec)과 감염에서 벗어나 회복된 이후에 얻은 샘플(Recovery)이 있다고 가정하자. 데이터 QC이후 UMAP까지 다 그린뒤, cell type annotation까지 마친 상태.
나는 이제 Infec 과 Recovery 사이에서 양적으로 차이를 보이는 cell type이 있는지 확인하고 싶다. 예를 들면, recovery에서 Monocyte의 수가 준다던가..
확률을 계산하기 위해선 먼저 cell type별로 그 비율을 계산해야겠지. 특정 Cell type이 Infec상태에서는 40%, Recovery에서는 20%의 비율로 나타났다고 치자. 그러면 각각의 Odds는
- Odd(Infec) = 0.4 / (1-0.4) = 0.67
- Odd(Recovery) = 0.2 / (1-0.2) = 0.25
- Odd ratio = 0.67 / 0.25 = 2.68
따라서 이 cell type은 Infec 상태에서 Recovery에 비해 약 2.86배 더 많이 나타난다고 할 수 있다.
이제 R 코드로 짜보자
Sample info.
clustering하고 cell type anootation까지 마쳤고, 난 celltype이 아니라 cluster단위로 condition에 따라 차이가 보이는지 궁금해서 그렇게 짜봄.
R code
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
# GEX.seurOBJ : 내 seurat object 이름
# 일단 Patient_cluster_condition 별로 cell 몇개인지 count
ncluster = length(levels(GEX.seurOBJ$wsnn_res.0.8))
Patient <- rep(c('P01','P02','P03','P04'),each=2*ncluster)
Condition <- rep(c("Infec","Recovery"), each=ncluster)
cluster <- levels(GEX.seurOBJ$wsnn_res.0.8)
data <- data.frame(Patient,Condition,cluster)
data$value <- 0
for(k in c('P01','P02','P03','P04')){
Psub <- subset(GEX.seurOBJ, subset=PatientID_re==k)
for(i in c("Infec","Recovery")){
Csub <- subset(Psub, subset=Condition==i)
total <- ncol(Csub)
for(j in levels(GEX.seurOBJ$wsnn_res.0.8)){
data[data$Patient==k & data$Condition==i & data$cluster==j,"value"] <- table(Csub$wsnn_res.0.8)[j]
}}}
# 각 환자별 전체 감염된 세포와 회복된 세포의 수를 계산
totals <- data %>%
group_by(Patient, Condition) %>%
summarise(total_count = sum(value)) %>%
pivot_wider(names_from = Condition, values_from = total_count, values_fill = list(total_count = 0))
# 각 환자와 클러스터별로 감염 및 회복 상태에서의 세포 수를 합산
data_ratio <- data %>%
group_by(Patient, cluster) %>%
summarise(infected_count = sum(value[Condition == "Infec"]),
recovery_count = sum(value[Condition == "Recovery"]),
.groups = 'drop') %>%
left_join(totals, by = "Patient") %>%
mutate(infected_ratio = infected_count / Infec,
recovery_ratio = recovery_count / Recovery,
infec_odd = if_else(infected_ratio == 0, 0, infected_ratio / (1 - infected_ratio)),
recovery_odd = if_else(recovery_ratio == 0, 0, recovery_ratio / (1 - recovery_ratio)),
odd_ratio = if_else(infec_odd == 0 | recovery_odd == 0, 0, infec_odd / recovery_odd)) %>% as.data.frame()
p_data<- data_ratio[,c(1,2,9,10)] %>% gather(key = "Condition", value = "Odd", infec_odd, recovery_odd) %>%
mutate(Condition = ifelse(Condition == "infec_odd", "Infec", "Recovery"))
p_data$cluster <- factor(p_data$cluster, levels=0:13)
# box plot
p <- ggplot(p_data, aes(x = factor(cluster), y = Odd)) +
geom_boxplot(aes(fill = Condition),width=0.5) +
geom_jitter(aes(color = Patient), width = 0.2) + # 환자별 점을 추가
scale_fill_manual(values = c("Recovery" = "blue", "Infec" = "red")) + # 색상 조정
labs(x = "Cluster", y = "Odds Ratio") +
theme_minimal()
# 통계적 유의성 검정 (예: Wilcoxon test) 및 p-value 추가
# 각 클러스터에 대해 별도로 유의성 검정을 수행하고 p-value를 얻습니다.
# 그리고 그 값을 그래프에 추가합니다.
for (cluster_level in unique(p_data$cluster)) {
# 클러스터별로 데이터 필터링
data_cluster <- p_data %>% filter(cluster == cluster_level)
# Wilcoxon test 수행
test_result <- wilcox.test(Odd ~ Condition, data = data_cluster)
print(paste0(cluster_level,":",test_result$p.value))
if (test_result$p.value < 0.05) {
max_y <- max(data_cluster$Odd, na.rm = TRUE)
p <- p + annotate("text", x = cluster_level, y = max_y, label = paste("p =", round(test_result$p.value, 3)),
size = 3, vjust = -0.5)
}
}
ggsave("OddRatio.png",width=12,height=4)
write.table(data_ratio, "CellTypeAnnot_OddRatio.txt", sep='\t')
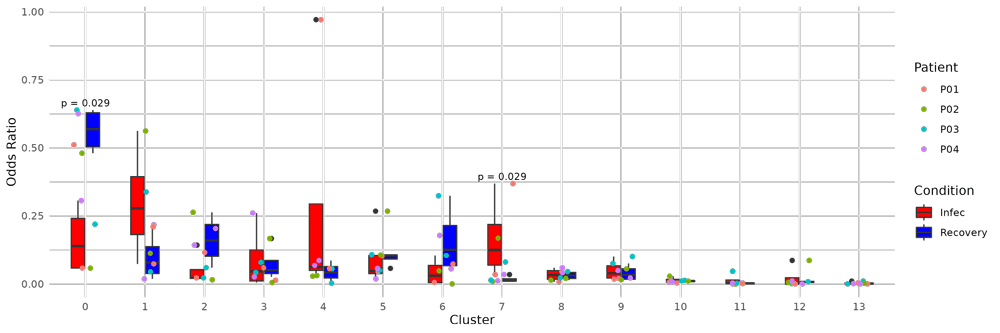
Result
환자가 4명이라 점이 4개밖에 안찍혀서 박스를 그리는 의미가 있나 싶지고 더 많았으면 좋겠지만ㅋㅋㅋㅋ
오늘도 하나 더 배웠다는데에 의의를 둔다. 데이터는 내잘못이 아니니까!
끝