[QC] DoubletFinder: 2024 업데이트 버전
이전포스트 및 관련 페이지
Mechanism & Processing, option관련 설명 이전 포스트 참조
👉DoubletFinder: doublet 제거법 01🔗github page
🔗publication link / Published: April 03, 2019 / DOI: https://doi.org/10.1016/j.cels.2019.03.003
Seurat v.5 지원
반년전쯤 DoubletFinder관련된 사용법을 정리할때까지만해도 Seurat v.5를 지원하지 않아서 따로 seurat v.3로 변환후 사용하는 방법을 사용했었는데, 얼마전에 (23년 11월) DoubletFinder 업데이트에서 Seurat v.5 호환이 가능하게 변경됐다! (Thanks for your update!)
이 업뎃에서 ‘_v3’ function들이 다 제거 돼서 나도 코드 업뎃하는겸 다시 포스팅하게 됨. + 이전에는 log-normalization을 사용했었는데 이번에는 single-cell 전용 툴인 SCTransform을 사용해서 진행해보려함.
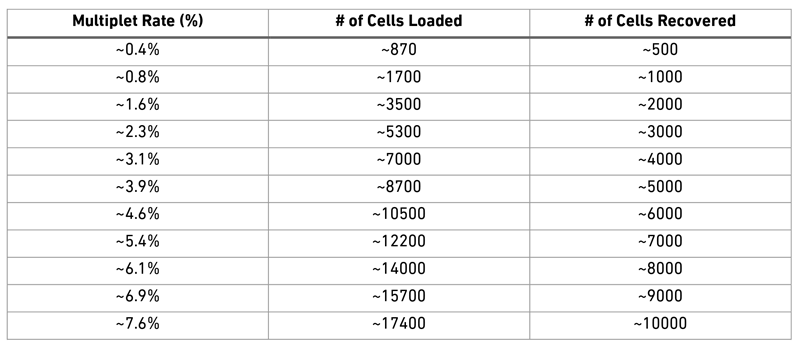
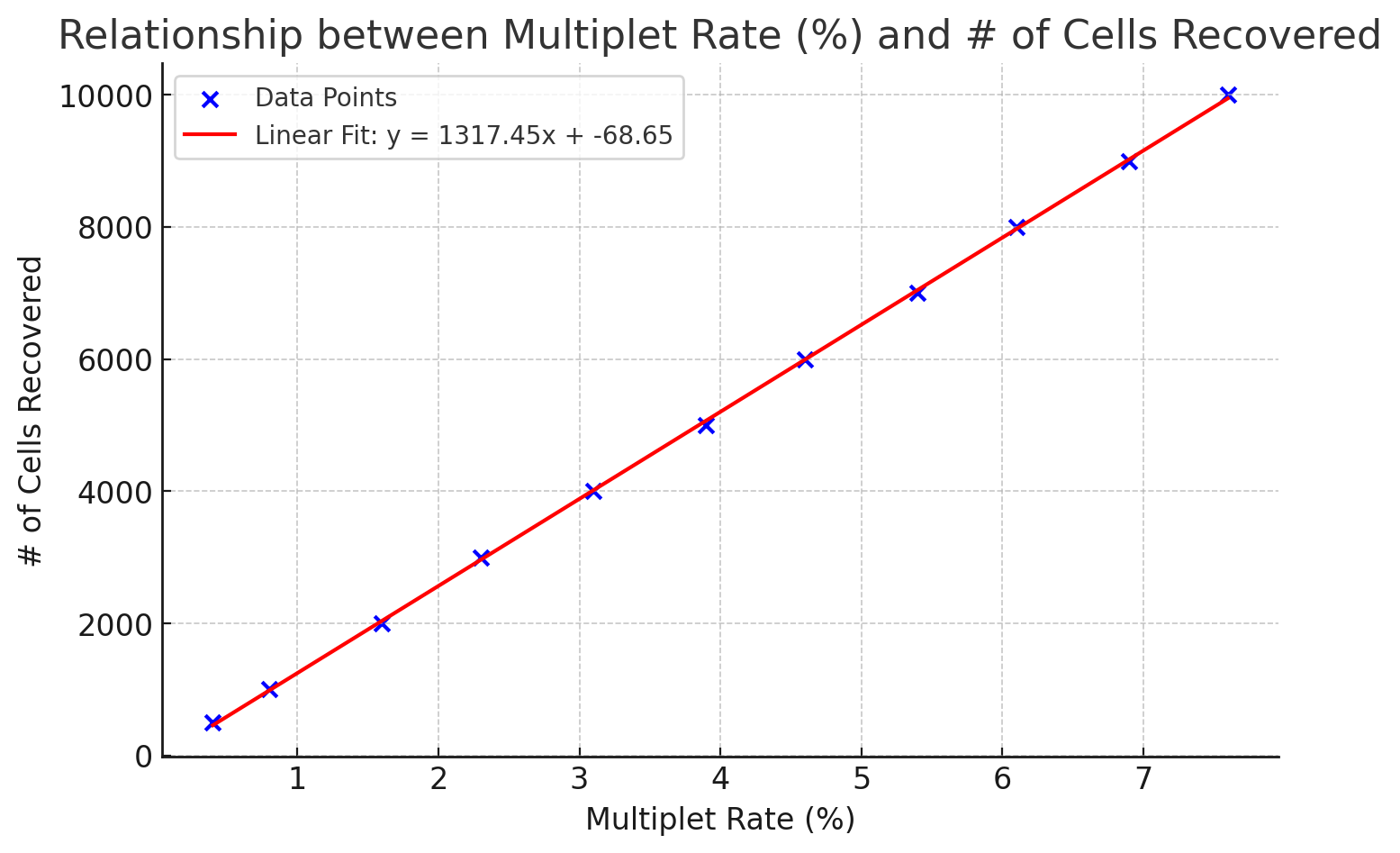
Multiplet rate 수식으로 변경
10X 홈페이지에서 제공하는 loading cell 수 별 multiplet rate 테이블을 chatGPT한테 주고 수식을 만들어 달라고 함.
Multiplet rate (%) = ((num of loaded cells) + 68.68 ) x 1317.45
Installation
1
2
3
4
remotes::install_github('chris-mcginnis-ucsf/DoubletFinder')
## for SCTransforom
BiocManager::install("glmGamPoi")
Usage
-
Seurat object- 있어야겠지, 난 QC이전에 CellRanger 결과 읽어들인 raw data(
paste0('00.',i,'_raw.rds'))를 준비했다. Seurat v.5
-
- sample 별 예상 dublet rate
- 이전 포스트 참고해서 시퀀싱이후 얻은 cell 갯수 대비 예상되는 doublet rate을 계상해서 준비 line 55
-
- paramSweep, doubletFinder
- 이전에는 뒤에 다
_v3붙었었는데 빼줌
-
- paramSweep(sct = TRUE)
- sct normalization 사용하고 싶으면 parameter설정해주기
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
library(Seurat)
options(Seurat.object.assay.version = "v5")
library(ggplot2)
library(patchwork)
library(tidyverse)
setwd('/your/working/dir')
SampleList <- c('S1','S2')
##############################################################################
## Doublet Finder
##############################################################################
library(DoubletFinder)
# load raw dataset
for(i in SampleList){
assign(i,readRDS(paste0('00.',i,'_raw.rds')))
}
for(i in SampleList){
file_name = paste0("DoubletFinder_",i,"_Doublet_cells.rds")
print(paste("Checking for file:", file_name)) # Debugging line
print(paste("File exists:", file.exists(file_name))) # Debugging line
if(file.exists(file_name)){
cat(paste("The files ", file_name, "exists. Skipping to next loop. \n"))
next
}
## Pre-process Seurat object (sctransform) -----------------------------------------------------------------------------------
seu <- get(i)
## Pre-process Seurat object (sctransform)
seu <- SCTransform(seu)
seu <- RunPCA(seu)
seu <- RunUMAP(seu, dims = 1:10)
seu <- FindNeighbors(seu, dims=1:10)
seu <- FindClusters(seu)
## pK Identification (no ground-truth) ---------------------------------------------------------------------------------------
sweep.res.list <- paramSweep(seu, PCs = 1:10, sct = TRUE)
sweep.stats <- summarizeSweep(sweep.res.list, GT = FALSE)
bcmvn <- find.pK(sweep.stats)
ggplot(bcmvn, aes(pK, BCmetric, group=1)) + geom_point() + geom_line()
ggsave(paste0("DoubletFinder_",i,"_peak.png"))
pK <- bcmvn %>% filter(BCmetric==max(BCmetric)) %>% select(pK)
pK <- as.numeric(as.character(pK[[1]]))
print(paste0(i,":",pK))
## Homotypic Doublet Proportion Estimate -------------------------------------------------------------------------------------
annotations <- seu$seurat_clusters
multiRate = ((ncol(seu)+68.65)/1317.45)/100 # 수식으로 계산한 multiplelet rate
homotypic.prop <- modelHomotypic(annotations)
nExp_poi <- round(multiRate*ncol(seu))
nExp_poi.adj <- round(nExp_poi*(1-homotypic.prop))
## Run DoubletFinder with varying classification stringencies ----------------------------------------------------------------
seu <- doubletFinder(seu, PCs = 1:10, pN = 0.25, pK = pK, nExp = nExp_poi.adj, reuse.pANN = FALSE, sct = TRUE)
DF.name = colnames(seu@meta.data)[grepl("DF.classification", colnames(seu@meta.data))]
DimPlot(seu, group.by = DF.name) + NoAxes()
ggsave(paste0("DoubletFinder_",i,".png"))
VlnPlot(seu, features = "nFeature_RNA", group.by = DF.name, pt.size = 0.1)
ggsave(paste0("DoubletFinder_",i,"_vln.png"),width=14)
saveRDS(colnames(seu[, seu@meta.data[, DF.name] == "Singlet"]),paste0("DoubletFinder_",i,"_Singlet_cells.rds"))
saveRDS(colnames(seu[, seu@meta.data[, DF.name] == "Doublet"]),paste0("DoubletFinder_",i,"_Doublet_cells.rds"))
}
끝
This post is licensed under
CC BY 4.0
by the author.