LINCS DB, perturbation data, 데이터 다운로드 및 R로 불러오기
LINCS: Library of Integrated Network-based Cellular Signatures
LINCS는 NIH의 지원을 받아 진행되고 있는 컨소시엄중 하나로, 여러 chemical, 유전적 변화(overexpression,knockout/down), 또는 질병 상태에 대한 세포의 다양한 반응을 조사해서 모아둔 데이터베이스다.
프로젝트의 주요 목표는 다양한 perturbing agent에 노출됐을때 세포들의 반응(유전자 발현 및 cellular process의 변화)을 광범위한 실험으로 데이터를 수집하고, 카테고리화해서 정리/보관함으로서 이를 통해 세포반응에 대한 network-based 데이터를 만들고 생물에 대한 이해를 높이는 것이다.
단순 데이터만 저장하고 있는게 아니라 visualization과 웹분석이 가능하도록 다양한 소프트웨어도 제공한다.
LINCS는 하나의 큰 프로젝트라고 하면 그 안에 다양한 실험 방법과 데이터 유형이 포함되어 있는데, RNA-seq, ATAC-seq, L1000 같은 실험 방법이 있다.
이 중 L1000은 LINCS 프로젝트의 일부로 개발된 특정한 기술로, 세포에서 일어나는 광범위한 유전자 발현 변화를 고속으로 측정하는 방법. L1000은 약 1000개의 대표 유전자를 대상으로 세포 내 유전자 발현의 변화를 측정한다. 이 1000개의 유전자는 세포 전체 유전자 발현 패턴을 추정하기 위한 대리 마커로 사용되는데, 전체 유전자를 다 분석하는것보다 적은 수의 유전자를 대상으로 함으로써, 실험 속도를 높이고 비용을 절감할 수 있었다.
Data Download
마침 우리학교 Ma’ayan lab이 이 컨소시움 소속으로 관련 연구를 하고 있어서 이 랩에서 개발한 SigCom이라는 디비를 이용했다. 정리를 잘해놨음.
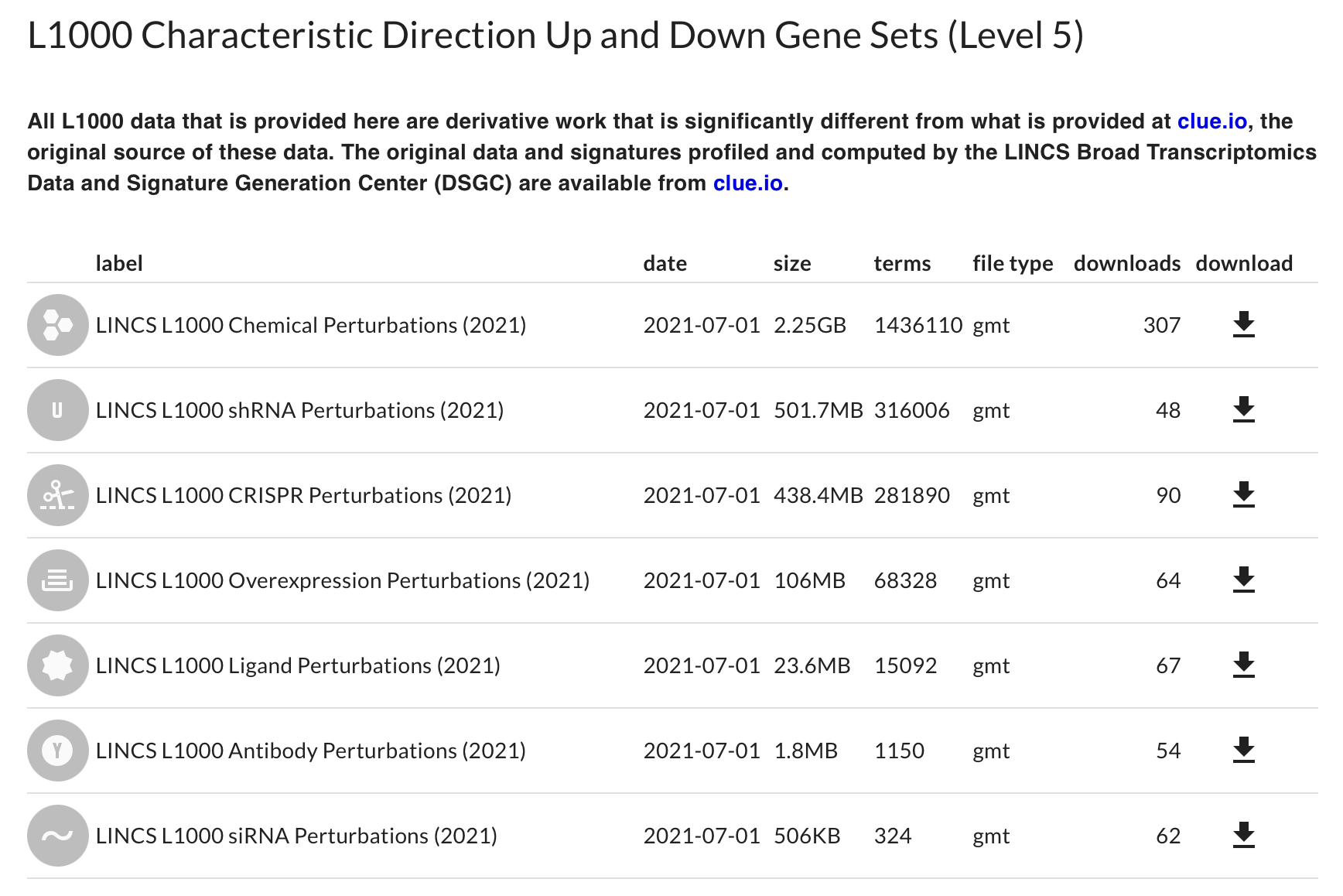
나는 Download tab에서 L1000 Characteristic Direction Up and Down Gene Sets (Level 5) 데이터를 다운 받았다.
1
2
3
4
wget https://lincs-dcic.s3.amazonaws.com/LINCS-sigs-2021/gmt/l1000_cp.gmt
wget https://lincs-dcic.s3.amazonaws.com/LINCS-sigs-2021/gmt/l1000_shRNA.gmt
wget https://lincs-dcic.s3.amazonaws.com/LINCS-sigs-2021/gmt/l1000_xpr.gmt
wget https://lincs-dcic.s3.amazonaws.com/LINCS-sigs-2021/gmt/l1000_oe.gmt
load in R
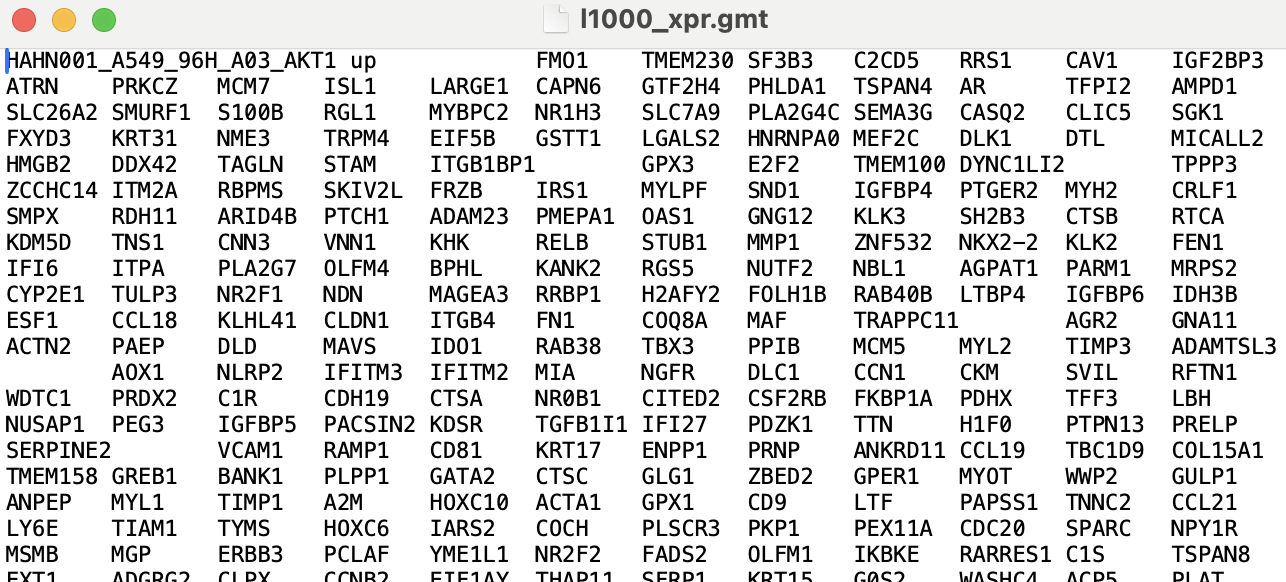
다운받은 파일은 대충 이런식으로 생겼다. 한줄에 perturbation title이랑 발현변화가 생긴 유전자 리스트.
읽어들이기 위한 function을 짜보자
1
2
3
4
5
6
7
read.geneSet.lincs <- function(geneSet.file){
gene.list <- readLines(geneSet.file)
gene.list <- strsplit(gene.list,"\t")
names(gene.list) <- sapply(gene.list,function(x) x[1])
gene.list <- lapply(gene.list,function(x) x[3:length(x)])
return(gene.list)}
1
lincs <- read.geneSet.lincs("l1000_xpr.gmt")
그러면 list형태로 읽어짐.